利用BioNumerics分析新冠病毒
一、数据的准备
1.参考基因序列(NCBI下载的Genbank文件)
2.待分析的序列(新冠病毒基因组数据)
3.存储毒株相关信息的Excel文件
二、创建新的数据库
点击数据库创建图标创建一个新的数据库,并对其命名。
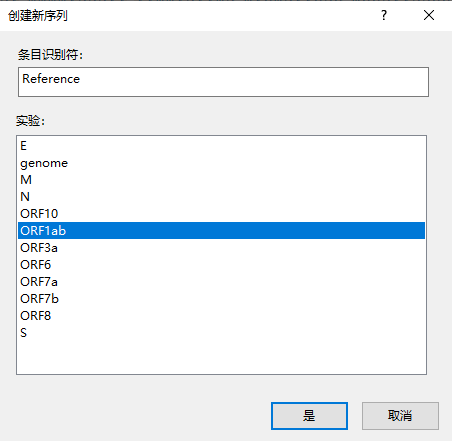
三、创建序列实验类型
创建数据库后进入到数据库主界面,点击实验类型创建图标
选择序列类型创建一个序列类型实验
对序列类型实验进行命名
点击完成后即创建好序列类型实验
同样的,按照上述方法根据CDS的名称依次创建序列类型实验
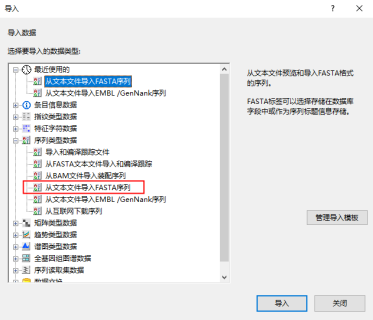
四、导入参考基因序列
点击数据库主界面左上角“文件”,选择“导入”
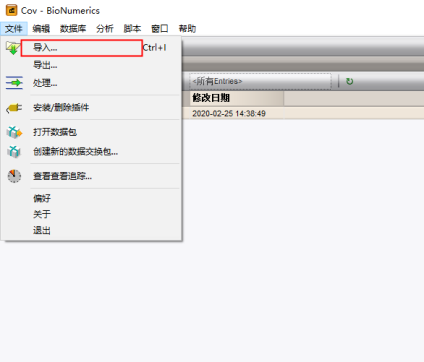
弹出的窗口中选择“序列类型数据”中的“从文本文件中导入EMBL /Genbank序列”
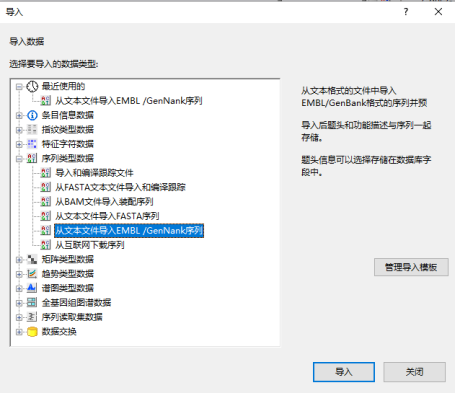
点击“浏览”选择参考基因序列文件
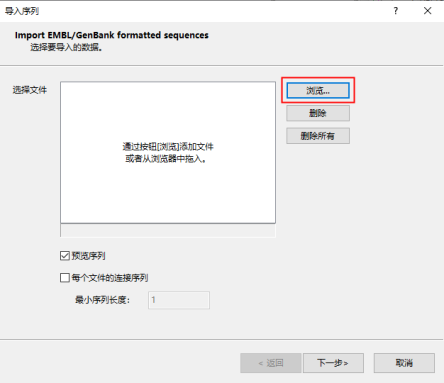
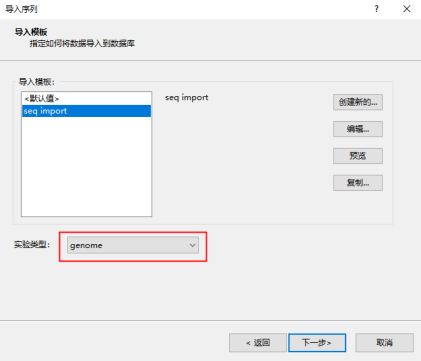
依次点击“下一步”
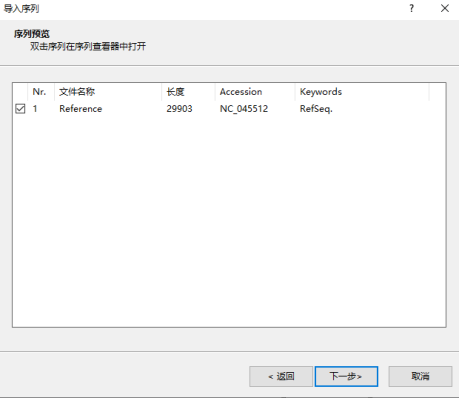
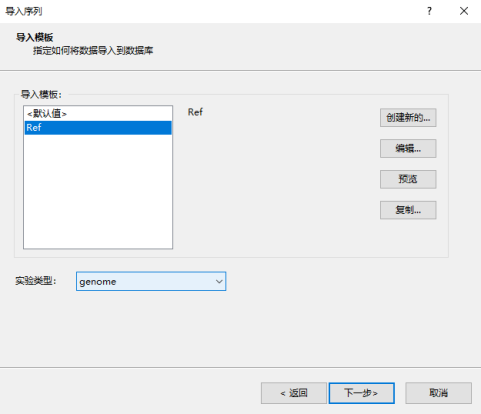
在导入模板窗口创建新的导入模板,注意此处实验类型应选择最初创建的序列实验genome
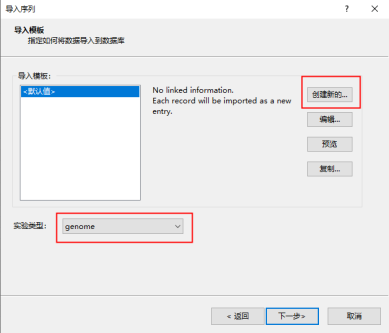
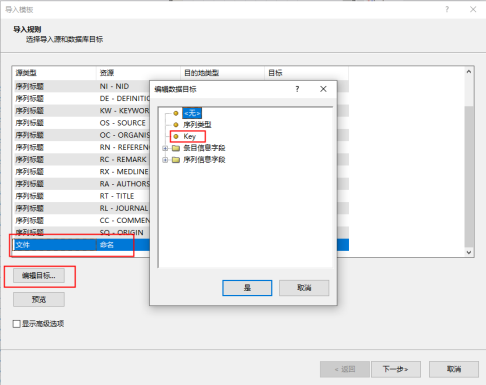
在导入规则窗口中选中源类型为“文件”一栏,然后点击编辑目标,和Key进行关联,再点击“是”,然后依次点击“下一步”、“完成”后对导入模板进行命名。
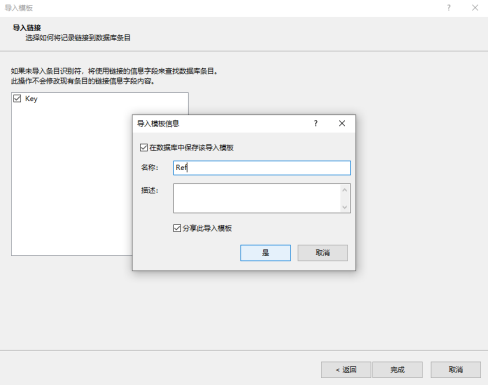
最后选择创建的导入模板依次点击“下一步”、“完成”后,参考序列即导入Bionumerics中
五、从参考序列中提取CDS序列
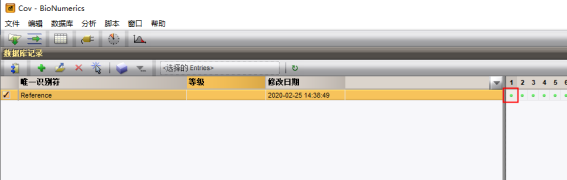
导入参考序列后,在数据库主界面的1号实验类型可以看到绿点,点击绿点则进入序列查看窗口
点击下方的CDS栏会选中CDS序列,右侧会显示对应CDS的名称
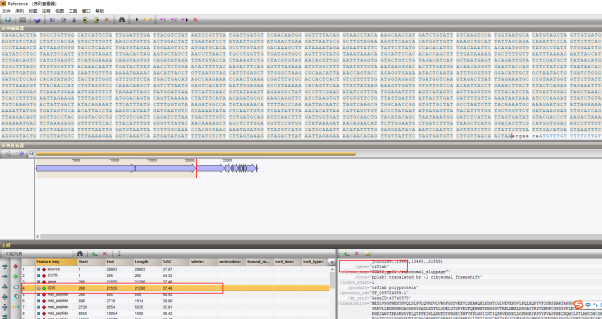
然后点击序列查看窗口左上角的“文件”,点击“保存选择”,在弹出的窗口中根据CDS的名称选择对应的实验。
按照上述方式,依次对其他CDS序列提取到相应的实验类型中
六、导入待分析的序列
和参考序列的导入类似,只是在导入时选择“序列类型数据”中的“从文本文件导入FASTA序列”。
此处支持序列文件的批量导入,后续的步骤和参考序列相同,都是创建新的导入模板,再按照模板导入序列文件。注意,此处的序列类型仍是选择“genome”
七、安装序列提取插件并设置
在数据库主界面点击左上角的“文件”,选择“安装/删除插件”,在弹出的窗口中的实用工具菜单选择“sequence extraction”插件并点击“Activate”进行激活
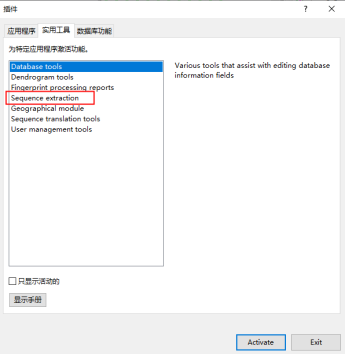
按照下图方式进入序列提取设置窗口
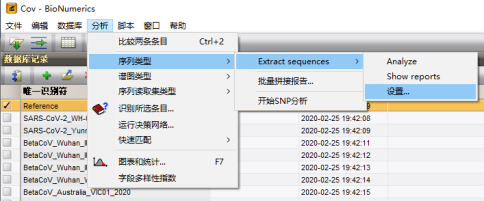
以“ORF1ab”为例,点击“添加”,按照下图进行选择
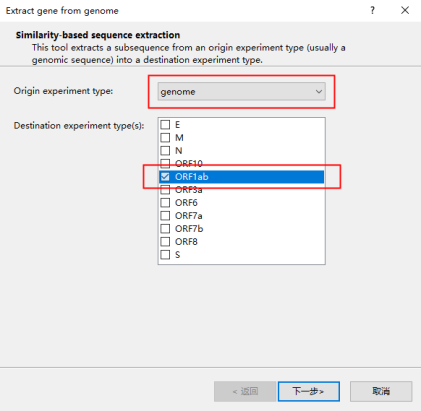
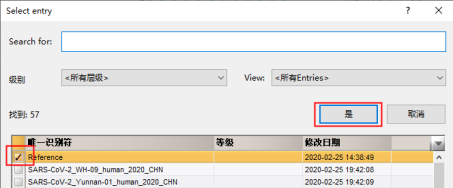
点击“下一步”后,点击“选择”,勾选参考序列条目后点击“是”
按照图中参数进行设置,点击“下一步”后,即设置完成,然后依次设置其他实验类型
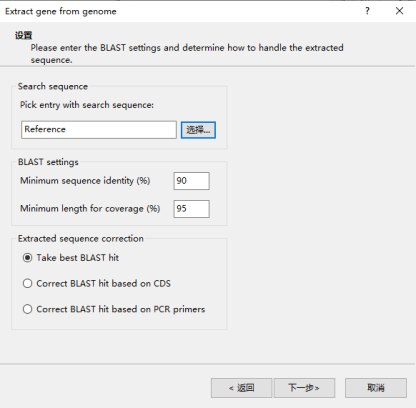
八、提取样品序列的CDS序列
在数据库主界面下,勾选所有样品序列,点击“分析”菜单,按照如图步骤进行分析
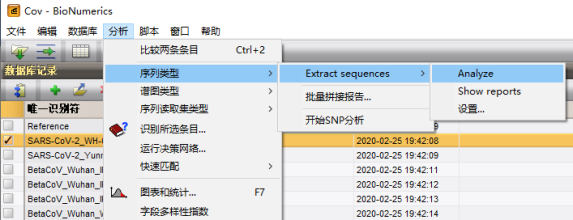
等待分析一段时间即可在其他序列实验看到提取的数据
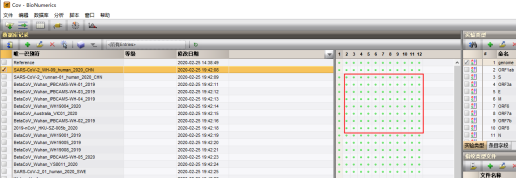
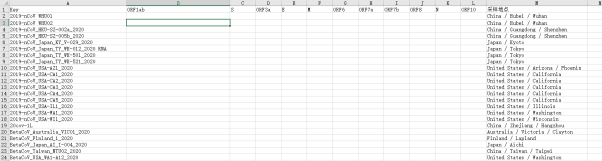
九、导入毒株信息
按照下图类似的Excel模板导入,CDS字段必需外,可扩展其他字段
点击数据导入图标,选择“条目信息数据”下的“导入字段(Excel文件)”,在导入模板界面,将key和key进行关联后,通过Shift键对其他字段进行全选,再点击“编辑目标”与“条目信息字段”进行关联
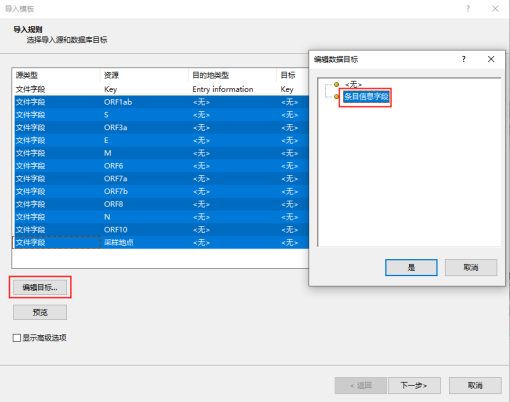
点击“是”后会弹出提示窗口,点击“是”即可
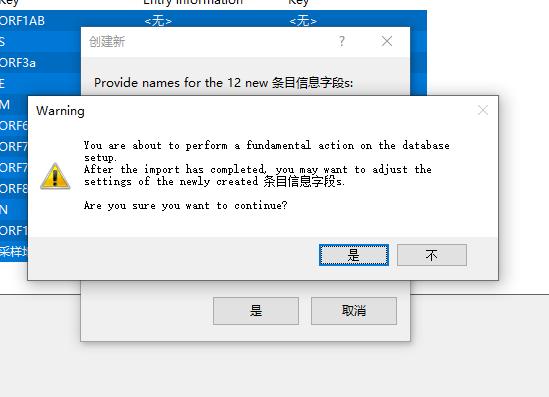
对导入模板命名后依次点击“下一步”、“完成”即可导入毒株信息
十、 在比对窗口进行序列比对
数据库主界面勾选所有样品序列后,点击右下方的比对,点击![]() 创建比对,此时弹出命名的窗口
创建比对,此时弹出命名的窗口
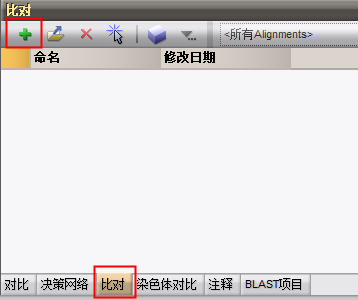
命名完后在弹出的窗口中选择CDS的序列实验,并点击“是”
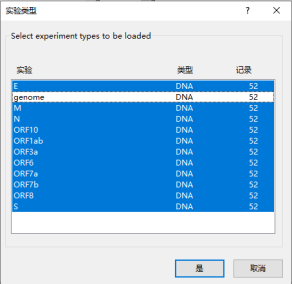
比对窗口内可以通过如图方式切换不同实验的序列比对
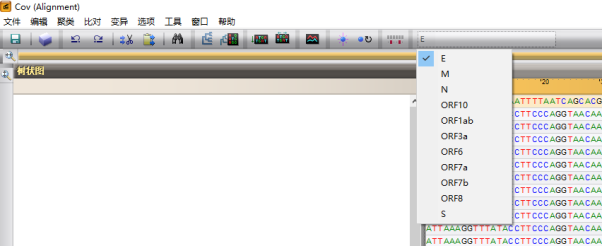
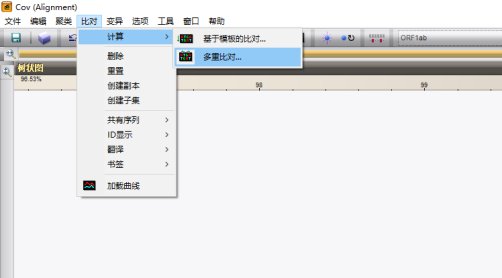
选择实验名称后按照下图方式进行多重序列比对
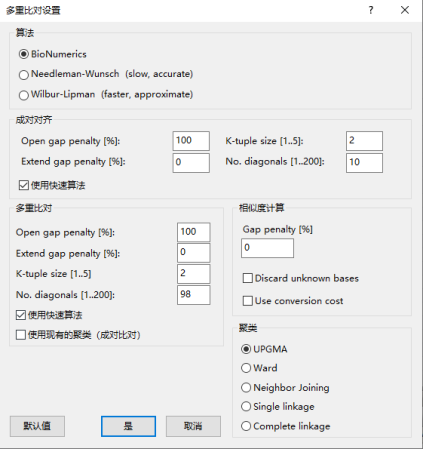
按照默认参数设置
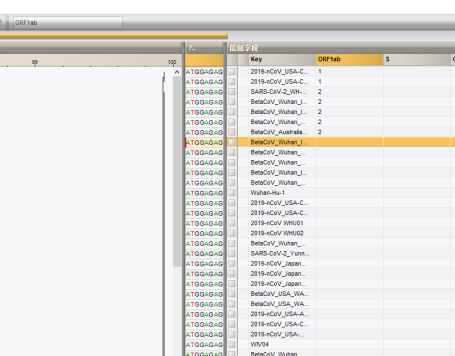
等待一段时间即出现序列的聚类图,根据结果按照MLST命名手动对相应的CDS进行编号
当同一编号的条目多时,可以勾选这些条目后回到数据库主界面,鼠标右键CDS名称的字段,批量进行命名,依次对所有序列实验比对并命名后即得到MLST结果
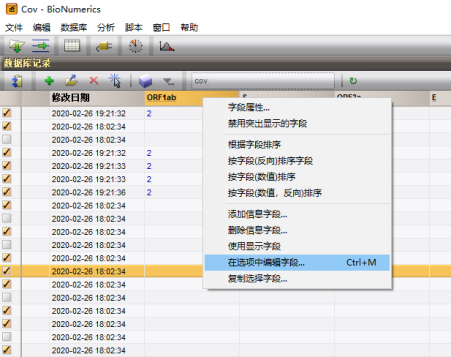
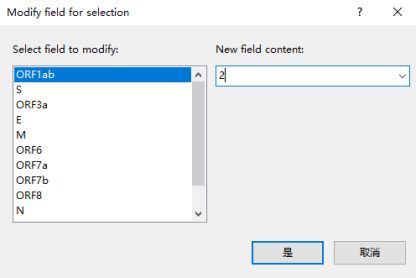
十一、导出MLST结果并导入Bionumerics分析
在数据库主界面按下图方式把结果保存至文件
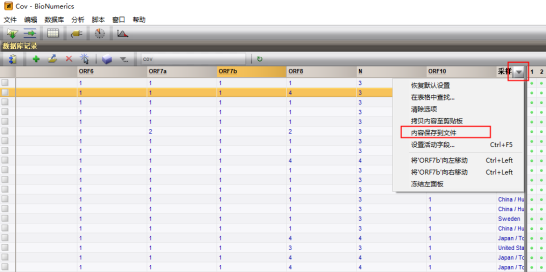
此时会生成Excel表格,将结果按照如下模板整理并另存为Excel文件
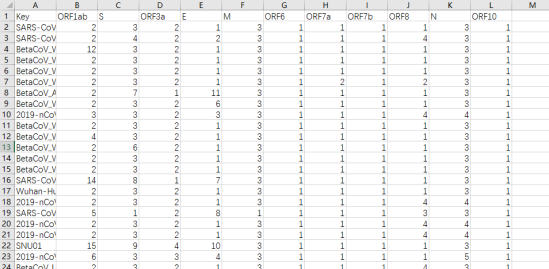
导入MLST结果前需要创建字符类型实验,依次按照默认设置创建即可
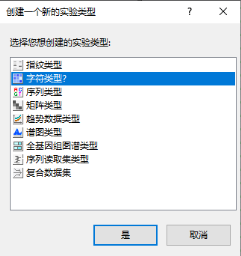
点击数据导入图标,选择“特征字符数据”下的“导入字段和字符(Excel文件)”
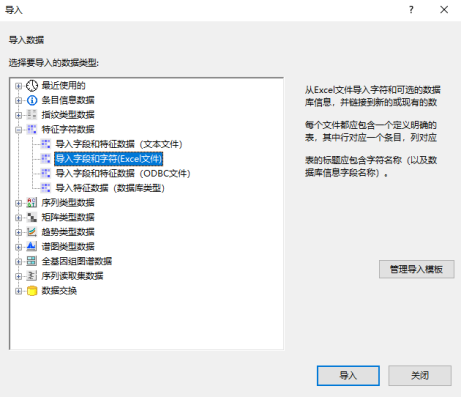
导入模板界面首先将Key和key进行关联,而各CDS结果是与特征值“MLST”关联。
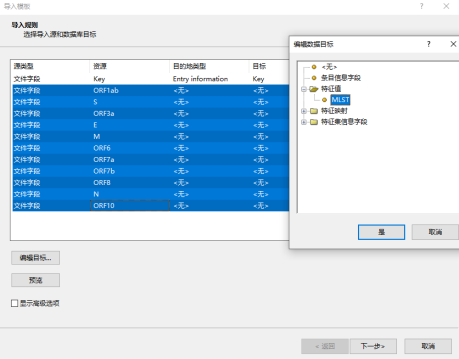
点击“是”后会弹出如下窗口,点击“是”即可
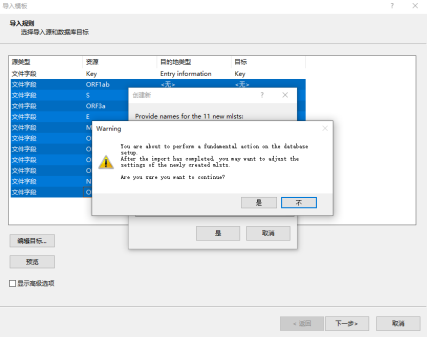
和其他数据导入类似,后续按照默认设置即可导入MLST结果
十二、MLST最小生成树构建
数据库主界面下勾选待分析的条目,创建对比
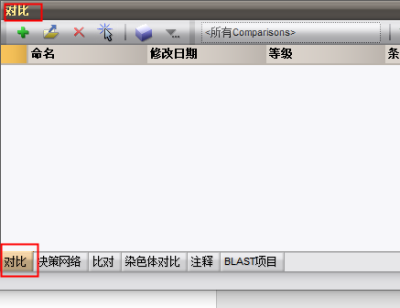
对比窗口点击MLST实验类型前的![]() 图标,并点击
图标,并点击![]() 显示值图标即可看到MLST结果
显示值图标即可看到MLST结果
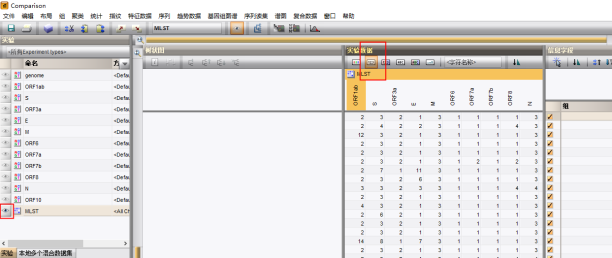
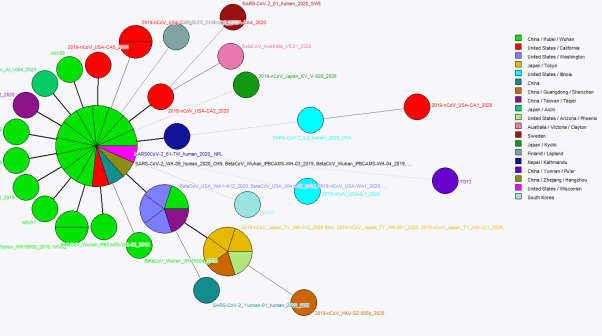
此时鼠标右键字段中的采样地点进行分组
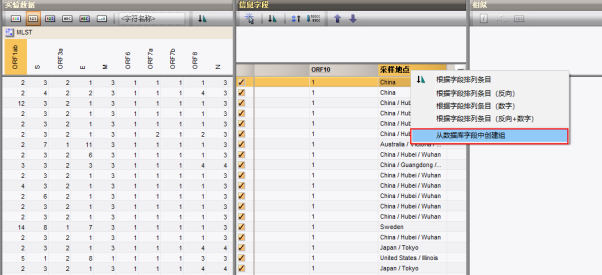
按照默认设置创建组后,条目即会以不同颜色进行分组
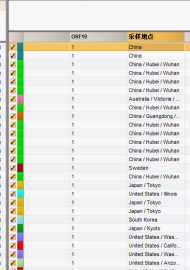
此时选择高级聚类分析
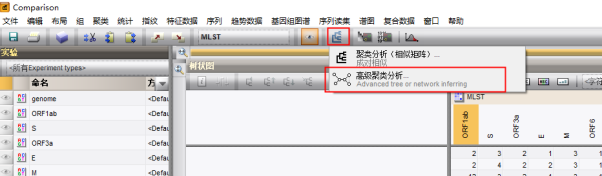
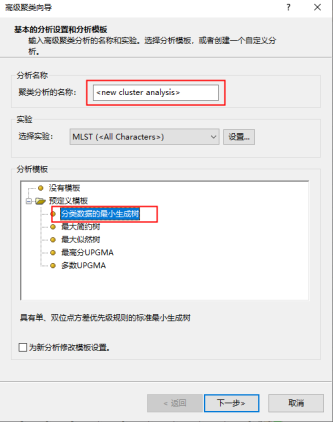
弹出的窗口中对聚类进行命名,并选择“分类数据的最小生成树”模板
点击“下一步”即生成MLST结果的最小生成树(Minimum Spanning Tree,MST),点击![]() 图标将图自动缩放显示
图标将图自动缩放显示
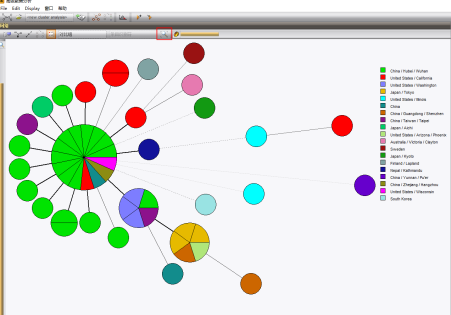
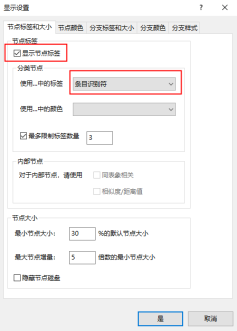
点击显示设置图标可以更改设置显示出条目的Key